南开科研团队在预测基因表达及解释非编码变异方面获◣重要进展
南开新闻网讯近日,我校软件学院讲师曾婉雯团队在预测基因表达及解释非编码变异方面获重要进展,研究成果以“Reusabilityreport:Compressingregulatorynetworkstovectorsforinterpretinggeneexpressionandgeneticvariants”为题,发表在Nature子刊《自然•机器智能》(NatureMachineIntelligence)上,期刊影响因子15.508。
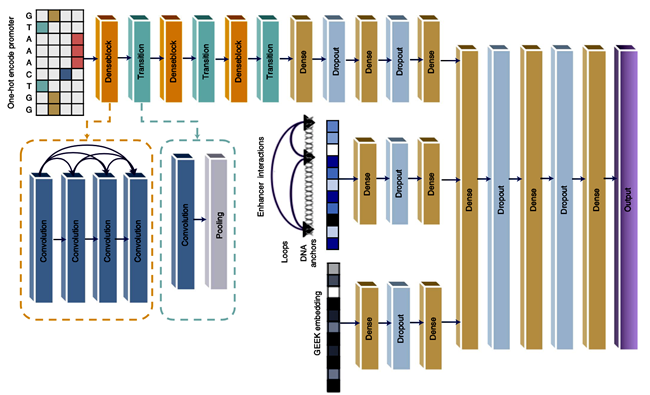
全基因组关联研究(GWAS)是确定复杂疾病风险基因↑/位ζ点的有效研究策略,为复ζ杂疾病的研究指明了方向,为实现个性化诊断、预后和治疗奠定了坚实基础,促进了人类遗传学和基因∞组学研究的发展。然而GWAS研究所得的大多数风险位点都位于非编码区,尚不清楚这些位点是否直接与疾病功能相关。而精准卐解释GWAS非编码区的位点是实现精准医疗的重要先决※条件。
单核苷酸多态性(SNPs)主要是指在基因组水平上由单个核苷酸↙的变异(碱基的转换或颠换、插入或缺失)所引起的DNA序列多态性。它是人类可遗传变异中最常见的⌒一种。大多数」的功能性非编码SNPs可以通过干」扰转录因子的结合和调控元件的功能来改变基因的表达,从而发挥其作用。值得注意的是㊣ ,这些调控元件具有高度的细胞类型特异性,这提示SNPs的功能性也有细胞类型特异性。因此需要在正确的组↓织和区域背景下对每一种细胞类型中活跃的调节元件进行分类和功能揭△示,结合其所形成的调控网络,进而帮助阐明常见神经退行性疾病分子发病机制中的①基因风险位点功能的重要性。
近年来,多组学技术尤其是表观染色质状态和三维结构测序技术得到了飞速发展与☉广泛应用,这对解析这些非编◆码区域变异的调控机制◆带来了新的契机。为系统解析调控网络对非编码遗传变异的影响,研究团队基于团队之前研◢发的DeepExpression模型,进一步整合序列数据、HiChIP三维基因组数据及GEEK模型调控网络数据的低维向▓量表示((lowdimensionalemedding),提高了对基因表达的预测能力。下游研究结果表卐明,整合序列数据、三维基因组数据和调控网络数据,能更好地理解转录调控机制∑ 和非编码变异的功能,为更精准解释GWAS遗传变异提供了新的方法。
南开大学软件学☆院为本文第一单位,南开大学软件学院讲师曾婉雯和斯坦福大学博士后信▼晶雪为共同第一作者,清华大学长聘副教授江瑞和中科院数学所王勇研究员为论文的共同通讯作者。该研究〖已得到国家自然科学基金青年项目资助、国家重点研发青年科学家项目资助。(软ω 件学院供稿)
论文链接:https://www.nature.com/articles/s42256-021-00371-6
未经允许不得转载:二九】年华大学门户 » 南开科研团队在预测基因表达及解释非编码变异方面获重要进展
- 上一篇:践行南开特色←研究性教学模式 推进“三一行动”计划落实
- 下一篇:南开助你“创”青春
相关推荐
- 【我和南开的故事】我有一段情
- 【组图】津南校区历史复建区:贯通历史 薪火相传
- 教育↑部党史学习教育高校第二指导组参加我校办公室、保密办党支部第一党小组专题组织生活会
- 我校参加全国教育系统疫情防控工作视频调度会议
- 南开学子在第◤十一届多语种全国口译大赛中获佳绩
- 魏大鹏率天津市政协委员代表团访问我校
- 南开大学研究团队在拓扑光子学领▆域再获新进展
- 南开学子在“新时代 新青年谈改革开放”高校媒体网评作品征集活动中获佳绩
- 郝亚明:种族歧视与仇外主义严重阻碍全ㄨ球疫情防控
- 发挥排球育人功能 践行南开体育精神
- 国务院应对新冠肺炎疫情联防联控机制综合组致信感№谢南开学者
- 南开大学召开2020年度校领导班子和领导干部考核及干部选拔任用“一报告两评议”工作会
- 南开大学2021新年音乐会在天津大◎剧院精彩上演
- 海南省教育厅厅长曹献坤一行来访我校
- 南开大学教师在第七届全国电工电子基础课¤程实验教学案例设计竞赛中获佳绩
- 电视剧《我们的西南联大》走进南开
- 南开大学分子微生物学与技术教育部重点实验室2020年度学术委员会召开
- 【迎百々年校庆】科技日报社总编辑刘亚东做客“百年南开大讲坛”
- 【迎百年校庆】南京大学张异宾教授◥做客“百年南开大讲坛”
- 南开大学召开党委常委(扩大)会暨中心组学习会
新闻公告
- 南开助你“创”青春 12-08
- 杨庆山检查学校安全工作 12-07
- 南开大学举办2021年度研究生指导教师培训活动 12-06
- 杨庆山会见校友企业家张文中一行 12-06
高考招生
- 南开大学2018年◥招生章程 08-05
- 南开大学2017年本科◆生招生章程 08-05
- 南开大学2016年本科生招〒生章程 08-05
- 南开大学2013年本科生招生章♀程 08-05
- 南开大学2014年本科生招生章程 08-05
- 南开大学2015年本科生招生章程 08-05
- 南開大學2013年招收香港地區免試生章♂程 08-05
- 南开大学2009年本科生招生章程 08-05
- 南开大学2011年本科生招生章程 08-05
- 南开大学2012年本科生招生章程 08-05
