我校学者研发出全基因组关联☆分析软件rMVP
南湖新〗闻网讯(通讯员 刘小磊)近日,我校动科动医学院赵书红教授团队研发了一款兼具计算≡高效、内存节省、可视化丰富的全基因组关联分析(Genome-Wide Association Study, GWAS)软件rMVP,其利用分块矩阵@ 计算、并行加速、计算流程优化等策略提升全基因组关联分析的计算效率,采用内存映射技术降低◣对内存资源的依赖,并提供灵活的基因组大数据处理与可视化模块,为大数据全≡基因组关联分析研究提供了新的工具。
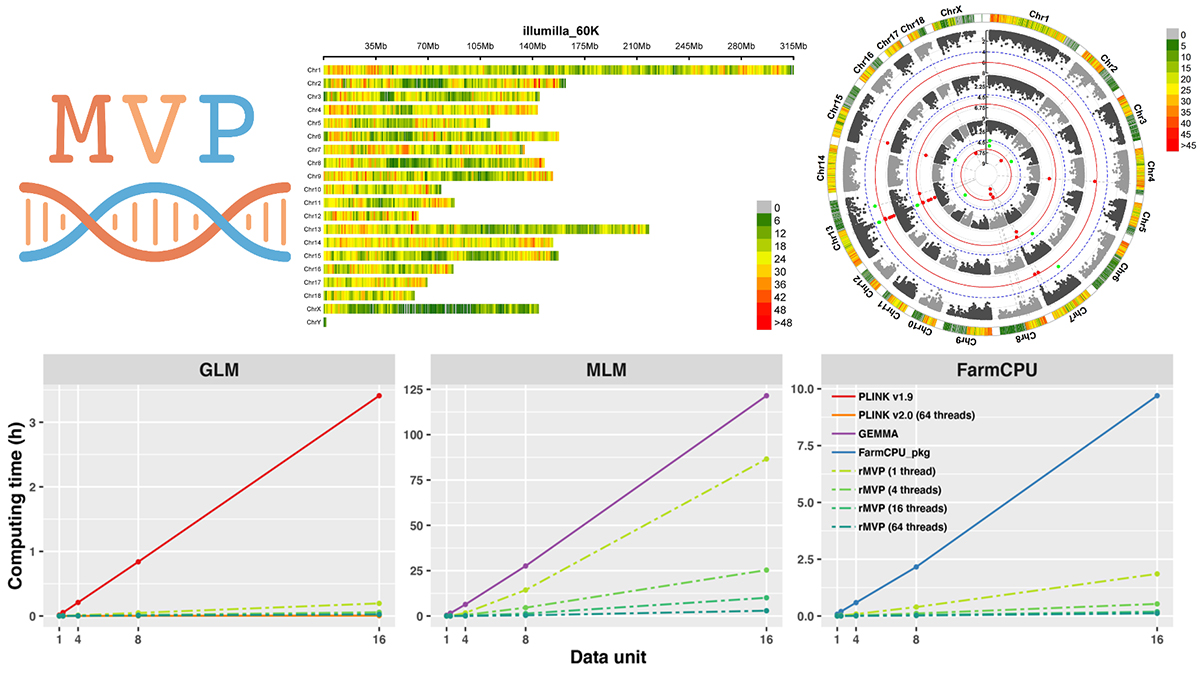
全基因组关联↘分析是一种通过统计学手段检验表型与覆盖全基因组的高密度标记之间关联程度来挖掘目标〇性状候选基因的重要方法。近十年来,GWAS被广泛应用于人类、畜牧、水产、植物和微生物等研究领域,鉴定了众多影响人类疾病与农业经济性状的关键基№因,成为解析复杂性状遗传机制的一把▃利器。随着测序成本的降低和表型组技术的发展,用于GWAS的群体数量、标记密度和表型数量迅速增长,尤其对于依赖个体亲缘▲关系矩阵的混合线性模型,其内存消耗呈现n2级增长(n为个体♀数量),庞大的数据规模给GWAS带来了新的ξ 挑战。因此,如何利用有限的计算资源高效地处理大数据成为目前GWAS领域的『研究重点之一。
rMVP软件兼具内存节省(Memory-efficient)、可视化丰富(Visualization-enhanced)、计算高效(Parallel-accelerated)等特点。通过内存映射技∮术,rMVP软件可直接从磁盘获取数值,有效︼降低了GWAS分析过程中的内存消耗,同时避免了多进程并行计算时的数据拷贝问〓题。它采用基于OpenMP技术和Intel MKL技术的“线程级”和“数据级”双并行模式进行计算加速,同时凭借分块矩阵拆分计算、GWAS流程全局优化等策←略避免了大矩阵的重复计◣算,这也使得rMVP比PLINK,GEMMA和FarmCPU_pkg中对应的模型计算速度快约5-20倍。
此外,rMVP软件还开发了一套高质量的可视化工具,可兼容 PLINK,GEMMA,GCTA,TASSEL等软件的分析结果,绘制包括表╱型分布图、标记密度分布图、群体结构◣图、曼哈顿图、QQ图在〒内的多种高分辨率图片。其中,它创新性地采用圆圈式曼哈顿图展示多性状、多模→型结果,并首㊣ 次将标记密度信息引入到曼哈顿图中。
本研究的共同第一作者为我校博士后尹立林、武汉卐理工大学博士生张浩浩,我校李新云教授和刘小磊副教授为共同通讯作者。
审核人:赵书红
【英文摘要】
Along with the development of high-throughput sequencing technologies, both sample size and SNP number are increasing rapidly in Genome-Wide Association Studies (GWAS), and the associated computation is more challenging than ever. Here, we present a Memory-efficient, Visualization-enhanced, and Parallel-accelerated R package called “rMVP” to address the need for improved GWAS computation. rMVP can 1) effectively process large GWAS data, 2) rapidly evaluate population structure, 3) efficiently estimate variance components by EMMAX, FaST-LMM, and HE regression algorithms, 4) implement parallel-accelerated association tests of markers using GLM, MLM, and FarmCPU methods, 5) compute fast with a globally efficient design in the GWAS processes, and 6) generate various visualizations of GWAS related information. Accelerated by block matrix multiplication strategy and multiple threads, the association test methods embedded in rMVP are approximately 5–20 times faster than PLINK, GEMMA, and FarmCPU_pkg. rMVP is freely available at https://github.com/xiaolei-lab/rMVP.
rMVP软件:https://github.com/xiaolei-lab/rMVP
论文链接:https://www.sciencedirect.com/science/article/pii/S1672022921000504
未经允许不得◆转载:二九╱年华大学门户 » 我校学者研发出全基因组关↙联分析软件rMVP
- 上一篇:我校召开十届纪委第∞九次全体(扩大)会议
- 下一篇:高翅调研指导新学期有关工作
相关推荐
- 关于做好个人防护∏的倡议
- 学校举行2019年冬季学位授予仪式
- 【战“疫”故事】“我们是战时物资保障员”
- 新生报到现场直击(一)
- 高翅、张启发赴深圳▲启动营养与健康研究院创建◤工作
- 【战“疫”故事】文法学院社会工作系将育人课堂开在战“疫”一线
- 吉林大学田毅鹏做客“狮子山社会学讲坛”
- 校党↓委围绕改革担当开展专题学习研讨
- 华中农业大学在动物源性成分检测方法上取得新进展
- 教育部副部长翁铁慧来校调∞研
- 中国疾病预防控制中心张永振教授畅谈“病毒”
- 本◥科生院赴湖北省部分优质生源基地开展招生宣传
- “祝鑫工作室”获批湖北省学生工作示☉范团队立项
- 学校举行“2021狮山欢乐节”筹备工作部署会
- 首期“外语论坛-文学讲堂”举行
- “时代楷模”朱有勇院士来◇校作报告
- 理学院58名师生共研新生大学第一堂课
- “决战脱贫攻坚 ,决胜全面小康”理论研讨会在学校召开
- 我校新增5名“享受政〖府特殊津贴”教授
- 4件作品获中国高校校报好新闻奖
新闻公告
- 高翅调研指导新学期有关工作 03-06
- 学校师生收看并热议十三届全国人大四次会议开幕会 03-05
- 我校学者研发出全基因组关联分析软件rMVP 03-04
- 我校召开十届纪委第九次全体(扩大)会议 03-04
- 导学共同体关键在于构建“志趣共同体” 03-04
- 导学共同体关键在于构建“志趣共同体” 03-04
- 桃花园里绘春色,师生融乐吟风雅 03-03
- 学校召开2021年第3、第4次党委常委会会议 03-02
- 校党政领导班子成员看望慰问新学期返校ω学生 03-01
高考招生
- 2017年华中农业大学普通本科招生章程 08-05
- 2018年华中农业大学普通本科招生章程 08-05
- 华中农业大学2015年全日制普通本科∴招生章程 08-05
- 华中农业大学普通本科招生章程(2016年) 08-05
- 华中农业大学2013年全日制普█通本科招生章程◆ 08-05
- 华中农业大学2014年全日制普通本科招生章〖程 08-05
- 华中农业大学2012年全日制普通本科招生章程 08-05
- 华中农业大学2008年全日制普通本科招生章程 08-05
- 华中农业大学2009年全日制普通本科招生章程 08-05
- 华中农业大学2007年全日制普通本科招生章程 08-05
